vcf具有表头部分和正文部分,其中表头部分是对正文部分中出现的缩写的解释。
vcf的正文部分,必须要有的是前面8列,一般来说有10列,分别是:
1 | CHROM |
CHROM 和 POS:参考序列名和variant的位置;如果是INDEL的话,位置是INDEL的第一个碱基位置。
ID:variant的ID。比如在dbSNP中有该SNP的id,则会在此行给出;若没有,则用”.”表示其为一个novel variant。
REF 和 ALT:参考序列的碱基 和 Variant的碱基。
QUAL:Phred格式(Phred_scaled)的质量值,表 示在该位点存在variant的可能性;该值越高,则variant的可能性越大;计算方法:Phred值 = -10 * log (1-p)。 p为variant存在的概率; 通过计算公式可以看出值为10的表示错误概率为0.1,该位点为variant的概率为90%。
FILTER:使用上一个QUAL值来进行过滤的话,是不够的。GATK能使用其它的方法来进行过滤,过滤结果中通过则该值为”PASS”;若variant不可靠,则该项不为”PASS”或”.”。
INFO:这一行是variant的详细信息,内容很多,以下再具体详述。
FORMAT 和 TTG11B:这两行合起来提供了’TTG11B′这个sample的基因型的信息。’TTG11B′代表这该名称的样品,是由BAM文件中的@RG下的 SM 标签决定的。
前面7列阐明该变异位点位于参考基因组的哪条染色体,哪个位置,是否被数据库给标记了ID(通常说的是dbSNP),该位置的参考基因组是什么碱基,这个变异位点变异成了什么碱基。找到这个变异的软件给它的质量值是多少,是否合格。
第8列 INFO 比较复杂,包含信息最多,看起来是一列,但是里面可以根据字段拆分成多列,都是 “TAG=Value”的形式,并使用”;”分隔。其中很多的TAG含义在VCF文件的头部注释信息##INFO中已给出。
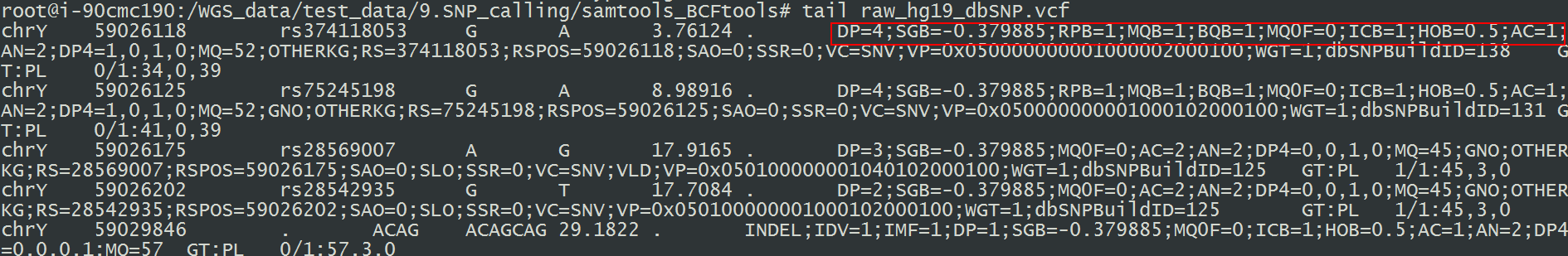
常见的TAG有:
AC,AF 和,AN[A开头的多和等位基因有关]:
AC(Allele Count) 表示该等位基因的数目;
AF(Allele Frequency) 表示等位基因的频率;
AN(Allele Number) 表示等位基因的总数目。
对于1个diploid sample[二倍体样本]而言:
基因型 0/1
表示sample为杂合子,等位基因数为1(双倍体的sample在该位点只有1个等位基因发生了突变),等位基因的频率为0.5(双倍体的
sample在该位点只有50%的等位基因发生了突变),总的等位基因为2;基因型 1/1
表示sample为纯合的,等位基因数为2,等位基因的频率为1,总的等位基因为2。
DP:reads覆盖度。是一些reads被过滤掉后的覆盖度。[注意,第八列和第九列都有DP,都表示该位点覆盖深度的信息,但是详细意义可能是不同的大家可以探究一下,在head里面就可以找到相应信息]
Dels:进行SNP和INDEL calling的结果中,有该TAG并且值为0表示该位点为SNV,没有则为INDEL。[可以根据这个tag分离indel和snv]
第9列信息:位点的基因型,测序深度的描述,一般有两列内容,前者为格式,后者为格式对应的数据。
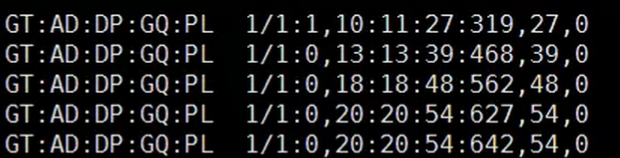
第九列包含标签有GT,DP,FT,GL,PL,GP等等,这些标签的含义可以在该vcf文件的表头里面找到。即vcf文件中以 ##FORMAT 开头的部分
GT:
样品的基因型(genotype)。两个数字中间用’/“分 开,这两个数字表示双倍体的sample的基因型。0表示样品中有ref的allele; 1 表示样品中variant的allele; 2表示有第二个variant的allele。
因此: 0/0表示sample中该位点为纯合的,和ref一致; 0/1 表示sample中该位点为杂合的,有ref和variant两个基因型; 1/1
表示sample中该位点为纯合的,和variant一致。
AD 和 DP:
AD(Allele Depth)为sample中每一种allele的reads覆盖度,在diploid中则是用逗号分割的两个值,前者对应ref基因型,后者对应variant基因型;
DP(Depth)为sample中该位点的覆盖度(一些reads被过滤掉的覆盖度)。
GQ:
基因型的质量值(Genotype Quality)。Phred格式(Phred_scaled)的质量值,表示在该位点该基因型存在的可能性;该值越高,则Genotype的可能性越大;计算方法:Phred值 = -10 * log (1-p) p为基因型存在的概率。
PL
指定三种基因型的质量值。这三种指定的基因型为(0/0,0/1,1/1),这三种基因型的概率总和为1。该值越大,表明为该种基因型的可能性越小。 Phred值 = -10 * log (p) p为基因型存在的概率。